[논문 정리] 8oxoG artifact
- 생물정보학/논문 정리
- 2020. 9. 5.

-목차-
1. Deep coverage targeted capture sequencing에서 FFPE 분석과 8oxoG artifact와의 관계
2. 8oxoG란?
3. 8oxoG가 생기는 원인
4. 8oxoG artifact를 감소시키는 방법
1. Deep coverage targeted capture sequencing에서 FFPE 분석과 8oxoG artifact와의 관계
1) DNA shearing: FFPE는 cfDNA와 달리 DNA shearing 단계를 거치고, 이때 8oxoG artifact가 발생하기 때문.
2) Low AF : low allele frequency에서 8oxoG(C>A) artifact가 많이 발생하며, 이는 somatic mutation과 구분이 어려워 True Positive의 somatic mutation 검출에 방해가 되기 때문.

2. 8oxoG란?
1) 8oxoG : 구아닌이 산화되어 8oxoG가 되며 이는 아데닌과 결합합니다. 그래서, G-C 염기쌍이 T-A 염기쌍으로 바뀌게되는 artifact를 의미합니다.
* 지칭: C>A/G>T transversion artifact라고 부르며, 짧게는 C>A artifact라고 부릅니다.

2) ArtQ score : 8oxoG의 정량화
*Definition : -10 x log10((consistent errors - inconsistent errors)/ all observations)
*설명: Phred score와 같은 컨셉입니다. ArtQ30 이면 해당 base가 8oxoG artifact가 아닐 확률이 99.9%이고, ArtQ20이면 해당 base가 8oxoG artifact가 아닐확률이 99%입니다.

3. 8oxoG가 생기는 원인
1) DNA shearing : shearing 이후 DNA 절편의 크기가 150bp일때와 500bp일 때의 비교입니다. 150bp일때 ArtQ score가 현저히 낮은걸 볼 수 있습니다.
Shearing 결과 base pair가 짧을 수록 더 높은 에너지가 필요하며, 더 높은 에너지를 쓸 수록 8oxoG가 더 많이 생긴다고 볼 수 있습니다.

2) Collection sites : DNA를 추출해주는 장소를 의미합니다.
논문에 따르면, 하나의 DNA 추출 장소에 대해 ArtQ score를 봤더니 전혀 패턴이 없었는데, 여러 DNA 추출 장소별로 ArtQ score를 봤더니 추출 장소별로 군집화되는 패턴이 생겼습니다.

4. 8oxoG artifact를 감소시키는 방법
1) 실험적 방법 : chelate 사용
chelate는 금속이온을 붙잡는 물질입니다. EDTA라는 킬레이트 사용시 NGS 결과 Library Yield도 적당히 나왔고, ArtQ score도 대조군(Tris-HCL alone)보다 높고, 다른 킬레이트에 비해서도 높은것을 확인 하였습니다.
단, DFAM은 ArtQ score가 가장 높은 킬레이트이지만, Library Yield가 하나도 나오지 않았습니다. 즉, 8oxoG artifact의 발생을 억제하는 척도도 중요하지만, NGS run 결과 Library Yield수치도 고려해야함을 알 수 있습니다.

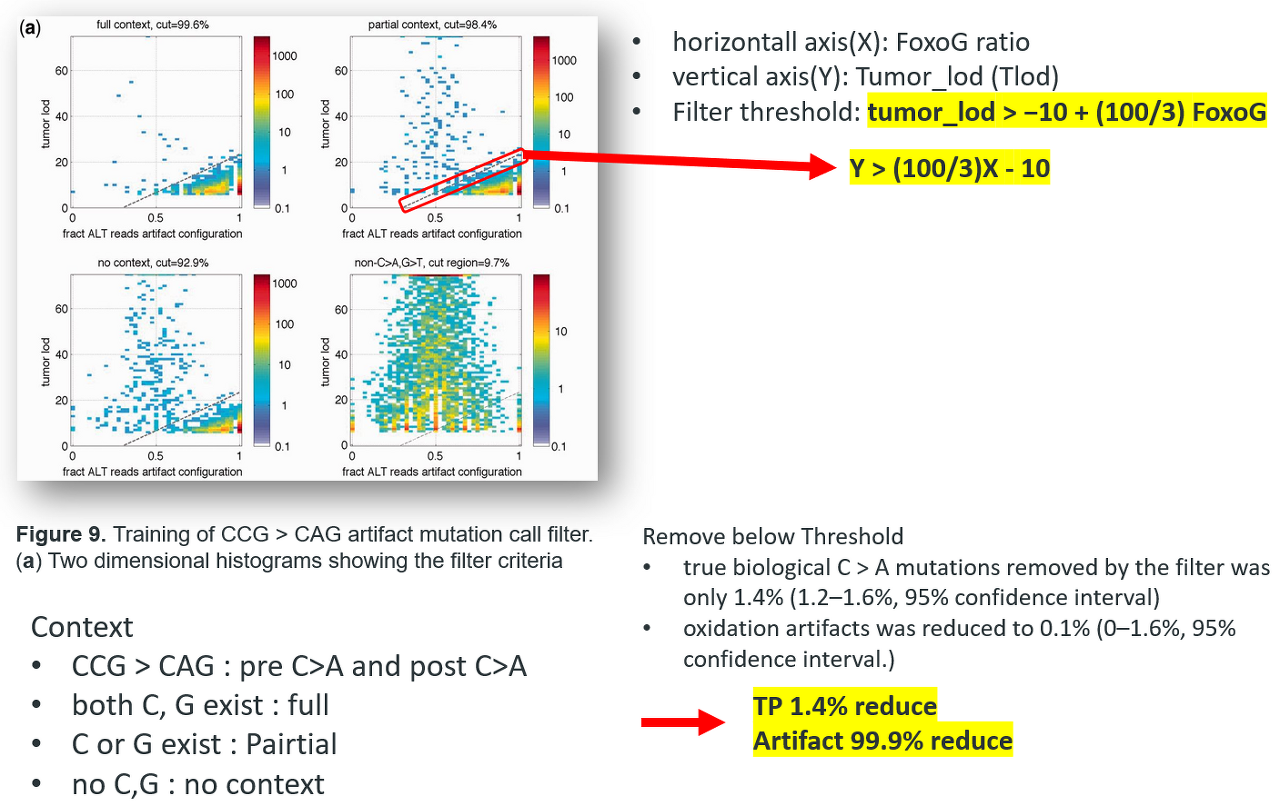
2) 분석적 방법 : tumor_lod와 FoxoG 수치 이용
* Definition
① tumor_lod : Tumor limit of Detection으로 somatic mutation으로 검출할 수 있는 Allele Frequency의 한계치를 의미합니다.
수식은 다음과 같습니다.

② FoxoG : Fraction oxoG의 줄임말입니다. 변이에 대해 oxoG의 비율을 의미합니다.
D-ToxoG (https://software.broadinstitute.org/cancer/cga/dtoxog)툴의 결과를 이용하면 구할 수 있는 수치입니다.

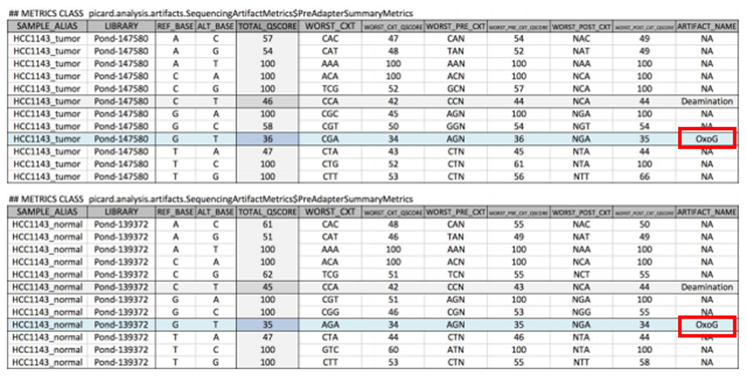
* Tumor_lod와 FoxoG를 이용한 8oxoG artifact Filtering
논문에선 Centext별로 나누어 Filtering을 진행하였습니다. Context란 변이에 대해 CCG > CAG 변이를 기준으로 하여 아래와 같이 Context를 구분합니다.
① full context : CCG > CAG[C>A 변이 앞, 뒤로 CG가 모두 존재] (Both C,G exist)
② partial context : CCG > CAG에서 C>A 변이를 기준으로 앞에 C or 뒤의 G중 하나만 존재하는 경우입니다. (C or G exist)
③ no context : CCG > CAG 에서 C>A 변이를 기준으로 앞에 C가 없고, 뒤에 G도 없는 경우입니다. (no C,G)
* Filtering은 Tumor_lod와 FoxoG 수치로 1차 부등식을 만들어 해당 영역에 존재하지 않는 변이들은 모두 Filter Out 하는 방식으로 진행했습니다.
Reference)
academic.oup.com/nar/article/41/6/e67/2902364
Discovery and characterization of artifactual mutations in deep coverage targeted capture sequencing data due to oxidative DNA d
Abstract. As researchers begin probing deep coverage sequencing data for increasingly rare mutations and subclonal events, the fidelity of next generation sequ
academic.oup.com
'생물정보학 > 논문 정리' 카테고리의 다른 글
| [논문 정리] Conpair - 샘플간 swap 확인 (1) | 2020.08.30 |
|---|