생물정보학 - Fragment size와 Insert size란?
- 생물정보학/생물정보학 이론
- 2020. 8. 29.
반응형
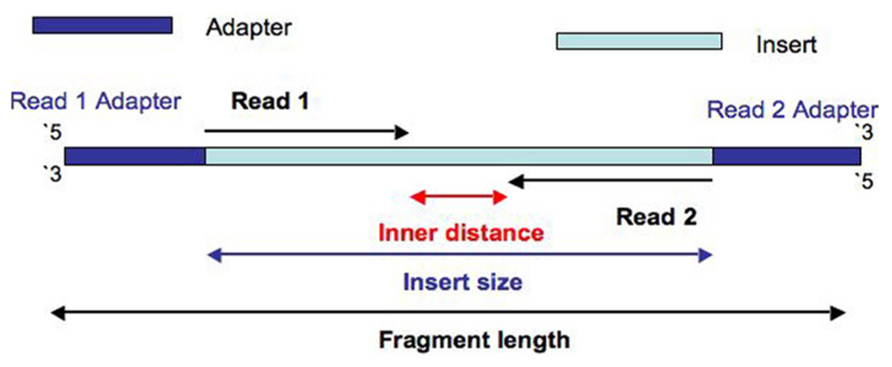
1. Fragment size와 Insert size란?
1) Fragment size(Fragment length): NGS 시퀀서에 들어간 실제 DNA 한가닥 길이. (Adapter sequence 길이 포함.)
2) Insert size: Adapter 사이에 '삽입된' DNA 길이(Insert). (Adapter제외한 DNA의 길이)
-> Adapter 사이의 거리를 의미. Inner mate distance라고도 함.
2. Fragment와 Read의 차이
1) Fragment: Adapter를 포함한 DNA.
2) Read: Adapter를 제외한 DNA.
즉, 위 그림에서 Insert를 방향에 따라 읽은 것을 Read1과 Read2로 나눈다.
- Read1 Read2 : NGS 장비에 들어가는 Paired End DNA는 양쪽 끝에서부터 DNA가 읽혀지기 때문에 왼쪽에서 오른쪽(5' to 3')으로 읽혀진 Read1과 오른쪽에서 왼쪽(3' to 5')으로 읽혀진 Read2가 있다.
Reference)
그림) Fragment size and Insert size : https://www.biostars.org/p/106291/
반응형
'생물정보학 > 생물정보학 이론' 카테고리의 다른 글
| VCF 파일 포맷: 변이 정보를 담은 포맷 (0) | 2020.08.31 |
|---|---|
| NGS - DNA 데이터 분석 한장 요약 (2) | 2020.08.26 |
| Rosalind란? 생물정보학 알고리즘 문제 풀기 (0) | 2020.08.19 |
| Bam/Sam 파일 포맷: 인간유전체 정보를 담은 포맷 (0) | 2020.08.18 |
| FASTQ 파일 포맷 : DNA 생(raw) 데이터 정보를 담은 포맷 (0) | 2020.08.04 |